

Orientador(a): Marcio Antonio Brunetto
Instituição: FMVZ USP
Trabalho classificado na 8ª Edição (2022) do Prêmio de Pesquisa PremieRpet®.
Índice
Resumo
O campo das ciências ômicas surgiu concomitante à ascensão da genômica, ciência que objetiva acessar e estudar o genótipo ou o fenótipo dos organismos. Por meio da química analítica e biologia molecular, é capaz de identificar genes-chave, quantificar a expressão gênica e analisar todos os metabólitos produzidos em resposta a fatores como ambiente, estado nutricional, estilo de vida, doenças ou intervenções terapêuticas. Portanto, esta revisão de literatura buscou descrever as diferentes metodologias e aplicações das ciências ômicas no estudo da biologia de sistemas, com foco na obesidade em cães como modelo.
Palavras-chave: Ciências Ômicas; Cães; Obesidade; Genômica; Transcriptômica; Proteômica; Metabolômica; Lipidômica
Introdução
As ciências ômicas têm como objetivo identificar, caracterizar e quantificar os componentes envolvidos nos mecanismos celulares de informação e expressão gênica, dos genes às proteínas sintetizadas e metabólitos circulantes. Desta forma, as ciências ômicas de célula única estudam os níveis intermediários entre o genótipo de um organismo e sua manifestação fenotípica, tendo como principais ramos de pesquisa a genômica, transcriptômica, proteômica e metabolômica (Aleksandrova et al., 2020).
A genômica investiga a sequência de nucleotídeos e genes codificantes para a expressão de determinados fenótipos, genes que serão traduzidos em RNAs, em especial o mRNA, que é amplamente estudado pela transcriptômica. O mRNA é transcrito em proteínas e estas participam de vias metabólicas em todo o organismo. Por meio de reações enzimáticas, geram diversos metabólitos que são objeto de estudo da metabolômica. A genômica e a transcriptômica têm sido utilizadas principalmente para a busca de genes-chave codificantes para determinada característica. Por outro lado, a proteômica e a metabolômica conseguem identificar e caracterizar uma grande quantidade de metabólitos em diferentes amostras. Estes diferentes focos das ciências ômicas, desde o estudo do genótipo até o fenótipo manifestado, estão sintetizados na Figura 1.
As ciências ômicas têm revelado aspectos importantes na compreensão biomolecular de doenças, pois estabelecem biomarcadores de importância para diagnósticos, definição de prognóstico, estratégias de prevenção e possibilitam o desenvolvimento de novas abordagens terapêuticas, uma vez que acessam as particularidades genéticas e metabólicas de cada indivíduo. Estas novas formas de tratamento são chamadas de “medicina personalizada” e consideradas um novo paradigma de compreensão de doenças complexas, uma vez que avaliam o background biomolecular do paciente para que a conduta terapêutica seja altamente individualizada e eficaz (Martorell-Marugán et al., 2019). Esta discussão é muito atual em medicina humana, porém ainda é pouco difundida na medicina veterinária. Portanto, por tratar-se de um tema complexo e pouco propagado, esta revisão de literatura busca esclarecer as diversas formas de aplicação das ciências ômicas em estudos da medicina veterinária e relatar as descobertas mais atuais.
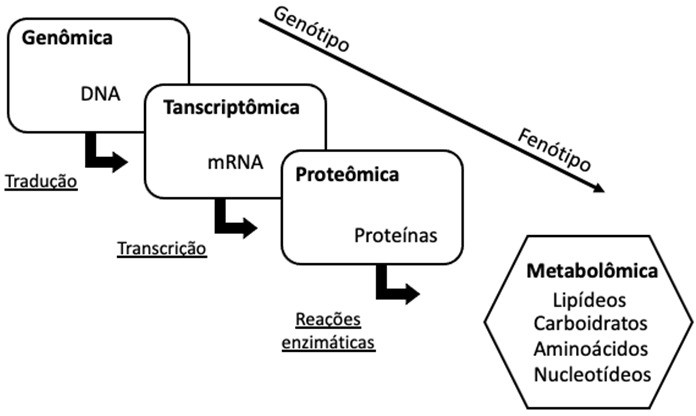
Genômica e transcriptômica
As pesquisas em genômica geralmente utilizam a Genome Wide-Linkage Studies (GWLS) e a Genome Wide-Association Studies (GWAS), metodologias que, respectivamente, comparam o código genético de uma linhagem de indivíduos e de toda a população. (Wallis & Raffan, 2020).
Tais técnicas possibilitam a compreensão das bases genéticas envolvidas no desenvolvimento de doenças como, por exemplo, a obesidade. Os principais marcadores genéticos para a obesidade canina descobertos a partir dessas técnicas foram uma deleção no gene POMC, codificante da propiomelanocortina (Raffan et al., 2016), maior frequência de polimorfismos no gene GPR, receptor acoplado à proteína G (Miyabe et al., 2015) e polimorfismos no gene CACNA1B (Sheet et al., 2020) e polimorfismos no gene TNF (Mankowska et al., 2020). O gene MC4R (receptor de melanocortina) foi pesquisado em diferentes raças de cães (Zeng & Zhang, 2014; Mankowska et al., 2017) e, apesar de ser um dos principais genes para a obesidade monogênica em humanos, não foi considerado um marcador para obesidade em cães.
Por outro lado, as tecnologias utilizadas na ciência transcriptômica incluem matrizes com base em sonda e, principalmente, sequenciamento de RNA. As análises transcriptômicas podem ter foco quantitativo, que avalia o nível de expressão gênica, ou qualitativo, por meio da detecção da presença de certas transcrições (Syed et al., 2019).
A expressão diferencial de genes específicos possibilita a caracterização genética de doenças crônicas e complexas como a obesidade canina. Por meio do sequenciamento de RNA em sangue de cães magros e obesos, Miyai et al. (2021) identificaram genes diferencialmente expressos no grupo dos cães obesos, quando comparado com os animais magros. Dentre os genes identificados, os autores destacaram: BCL2L15, LOC100855540, ALAS2 e FOLH1. O gene BCL2L15 teve maior expressão no grupo obeso, enquanto os outros genes citados demonstraram expressão reduzida. O gene BCL2L15 está associado à regulação de apoptose celular pelo controle da permeabilidade de membrana da mitocôndria, de forma que maior expressão desse gene pode estar associada ao aumento da apoptose celular. O gene FOLH em expressão diminuída está relacionado à menor absorção de folatos por meio da codificação de enzimas que hidrolisam poliglutamil-folatos. Ademais, os genes LOC100855540 e ALAS2 estão envolvidos na codificação da subunidade alfa da hemoglobina e na diferenciação de eritrócitos, cujas expressões alteradas afetam o metabolismo energético mitocondrial (Miyai et al., 2021).
Proteômica e metabolômica
A proteômica é a ciência ômica que caracteriza e quantifica as proteínas celulares, e suas vantagens são detectar modificações pós-transducionais de proteínas, bem como construir ligações funcionais entre os compostos proteicos. A principal metodologia é a Cromatografia Líquida com Espectrometria de Massa (LC-MS), método também utilizado em análises metabolômicas. Uma das principais vantagens da proteômica reside no fato de poder utilizar a saliva como amostra para os estudos (Torres et al., 2018; Lucena et al., 2020).
Um catálogo do proteoma salivar de cães saudáveis foi criado por Torres et al. (2018), o que possibilita comparações entre diferentes status de doença nesta espécie. Dessa forma, Lucena et al. (2020) realizaram comparações entre a proteômica salivar de cães da raça Beagle obesos e após a perda de peso. Das 248 proteínas salivares quantificadas, 23 apresentaram diferença entre os grupos experimentais. Os autores apontaram que tais proteínas estariam relacionadas ao sistema imunológico, estado inflamatório, estresse oxidativo e metabolismo da glicose (Lucena et al., 2020).
A caracterização dos metabólitos produzidos por um organismo é uma das formas mais atuais de acessar seu fenótipo, pois estes representam os produtos da expressão gênica e ações de proteínas (Carlos et al., 2020) e, por isso, fornecem informações precisas sobre o fenótipo de um indivíduo em determinado tempo. Por sua alta especificidade aos metabólitos lipídicos, a lipidômica surge como um ramo da metabolômica de interesse para o estudo nutricional dos sistemas biológicos (Avela & Sirén, 2020).
A lipidômica plasmática pode distinguir diferentes raças caninas mesmo em ambientes e dietas não padronizadas (Boretti et al., 2020) e o perfil lipídico sérico também difere entre cães de raça e ambientes padronizados recebendo diferentes dietas (Lloyd et al., 2017). Estes últimos autores avaliaram diferentes grupos de cães que foram manejados com alimento seco extrusado e dieta caseira crua suplementada com óleo de linhaça, ambas completas e balanceadas. Os animais alimentados com a dieta caseira apresentaram maior quantidade de frações lipídicas contendo ácidos graxos ômega-3, mas as frações saturadas, mono e di-insaturadas estavam em menores concentrações neste grupo. Também foi observado aumento significativo em uma molécula da classe das esfingomielinas, a esfingosina-1-fosfato (S1P), envolvida em processos de sinalização de reações imunes, integridade vascular e apoptose (Lloyd et al., 2017).
O estudo de Hoffman et al. (2021) avaliou as alterações no metabolismo de lipídeos em cães de diferentes pesos corporais e idades, e buscou investigar as diferenças fisiológicas que explicam porque diferentes portes de cães têm longevidades tão distintas. Os resultados sugeriram que cães de maior porte, cuja expectativa de vida é menor, têm maior concentração de esfingomielinas, enquanto triglicerídeos estão aumentados em cães de maior idade, independente do porte.
Estudos envolvendo lipidômica em cães são recentes e a literatura ainda é escassa, evidenciando a necessidade de maior investigação neste campo, especialmente no que tange à obesidade. Até o momento, apenas um estudo se propôs a identificar, por meio da lipidômica, as alterações metabólicas consequentes à obesidade (Söder et al., 2019). Estes autores apontaram “inflexibilidade metabólica” (termo utilizado no estudo) à ingestão alimentar em cães da raça Labrador Retriever em sobrepeso crônico, ou seja, com escore de condição corporal (ECC) acima de 6 (Laflamme, 1997). Neste estudo, cães em situação de obesidade ou sobrepeso apresentaram menores concentrações séricas basais de carnitina e acetil-carnitina, moléculas responsáveis pelo transporte de ácidos-graxos para a mitocôndria. Por outro lado, no grupo de cães em ECC ideal, houve diminuição da concentração de acetil-carnitina e carnitina séricas apenas após a alimentação, indicando “flexibilidade metabólica” do organismo na obtenção de alimento como principal fonte de energia em detrimento dos estoques corporais. Não houve alteração neste perfil metabólico dos cães obesos após a alimentação (Söder et al. 2019). Cães em leve sobrepeso já apresentavam a “inflexibilidade metabólica” observada no perfil de cães obesos. Por conta disso, os autores hipotetizaram que a “inflexibilidade metabólica” pode ser influenciada por outros fatores além da obesidade e que, uma vez adquirida, pode ser um fator para o desenvolvimento da condição obesa.
Considerações Finais
O conhecimento sobre as bases genéticas, vias metabólicas e os mecanismos subjacentes ao desenvolvimento dos diferentes distúrbios nutricionais e impactos de diferentes dietas pode ser amplamente complementado pelas ciências ômicas supracitadas, visto que este campo de estudos trabalha com grande quantidade de informações sobre um indivíduo. Tais metodologias são capazes de identificar moléculas e permitir a caracterização de vias metabólicas com profundidade e especificidade não alcançadas por outras tecnologias.
Referências bibliográficas
ALEKSANDROVA, K. et al. Omics Biomarkers in Obesity: Novel Etiological Insights and Targets for Precision Prevention. Current Obesity Reports, v. 9, n. 3, 219–230, 27 set. 2020.
AVELA, H. F.; SIRÉN, H. Advances in lipidomics. Clinica Chimica Acta. V. 510, p. 123-141, 2020.
HOFFMAN, J. M. et al. Alterations of Lipid Metabolism With Age and Weight in Companion Dogs. The Journals of Gerontology: Series A. V. 73, n. 3, p. 400-405, 2021.
LAFLAMME, D. Development and validation of a body condition score system for dogs. Canine Practice, 22, 10-15, 1997.
LUCENA, S. et al. Changes in the salivary proteome of beagle dogs after weight loss. Domestic Animal Endocrinology, v. 72, p. 106474, 1 jul. 2020b.
MANKOWSKA, M. et al. Sequence analysis of three canine adipokine genes revealed an association between TNF polymorphisms and obesity in Labrador dogs. Animal Genetics, v. 47, n. 2, p. 245–249, abr. 2016.
MANKOWSKA, M. et al. Polymorphism and methylation of the MC4R gene in obese and non-obese dogs. Molecular Biology Reports, v. 44, n. 4, p. 333–339, 28 ago. 2017.
MARTORELL-MARUGÁN, J. et al. Deep Learning in Omics Data Analysis and Precision Medicine. In: Computational Biology. [s.l.] Codon Publications, 2019. 37–53.
MIYABE, M. et al. Genetic variants of the unsaturated fatty acid receptor GPR120 relating to obesity in dogs. Journal of Veterinary Medical Science, v. 77, n. 10, 1201–1206, 2015.
RANGEL-HUERTA, O. D.; PASTOR-VILLAESCUSA, B.; GIL, A. Are we close to defining a metabolomic signature of human obesity? A systematic review of metabolomics studies. Metabolomics, v. 15, n. 6, p. 93, 13 jun. 2019.
RAFFAN, E. et al. A Deletion in the Canine POMC Gene Is Associated with Weight and Appetite in Obesity-Prone Labrador Retriever Dogs. Cell Metabolism, v. 23, 5, p. 893–900, maio 2016a.
SHEET, S. et al. Identification of Candidate Genes and Pathways Associated with Obesity-Related Traits in Canines via Gene-Set Enrichment and Pathway-Based GWAS Analysis. Animals, v. 10, n. 11, p. 2071, 9 nov. 2020.
SÖDER, J. et al. Indication of metabolic inflexibility to food intake in spontaneously overweight Labrador Retriever dogs. BMC Veterinary Research, v. 15, n. 1, p. 96, 20 dez. 2019.
SYED, N. A. et al. Single-Cell Omics in Metabolic Disorders. In: Single-Cell Omics. [s.l.] Elsevier, 2019. p. 153–164.
TORRES, S. M. F. et al. Salivary proteomics of healthy dogs: An in depth catalog. PLOS ONE, v. 13, n. 1, 201
WALLIS, N.; RAFFAN, E. The Genetic Basis of Obesity and Related Metabolic Diseases in Humans and Companion Animals. Genes, v. 11, n. 11, p. 1378, 20 nov. 2020.
ZENG, R.; ZHANG, Y.; DU, P. The SNPs of Melanocortin 4 Receptor (MC4R) Associated with Body Weight in Beagle Dogs. Experimental Animals, v. 63, n. 1, 73–78, 2014.





